Séance big data#
Links: notebook, html, PDF, python, slides, GitHub
Découverte de plusieurs façons de calculer des statistiques sur des fichiers volumineux.
from jyquickhelper import add_notebook_menu
add_notebook_menu()
%matplotlib inline
Les données#
On récupère un fichier de la DAMIR. Le fichier une fois décompressé fait plus de quatre Go. Vous pouvez le télécharger directement depuis le site ou prendre une version de backup. Vous trouvez d’autres données intéressantes sur le sujet sur le projet SGMAP-AGD/DAMIR.
import os
filename = "bigdata/A201612_small.csv"
# pour travailler avec un fichier plus gros (4 Go)
# filename = "bigdata/A201612.csv"
xlsfile = "bigdata/Lexique_open-DAMIR.xls"
if not os.path.exists(filename):
from pyensae.datasource import download_data
if not os.path.exists("bigdata"):
os.mkdir("bigdata")
filenamegz = filename.replace(".csv", ".csv.gz")
download_data(os.path.split(filenamegz)[-1], whereTo=".")
download_data(os.path.split(xlsfile)[-1], whereTo="bigdata")
if not os.path.exists(filename):
filename = os.path.split(filename)[-1]
os.stat(filename).st_size / 2**30
0.015162252821028233
Il est impossible de le charger en mémoire en entier. On regarde les premières lignes.
with open(filename, "r") as f:
for i, line in enumerate(f):
print(line.strip("\r\n"))
if i >= 5:
break
FLX_ANN_MOI;ORG_CLE_REG;AGE_BEN_SNDS;BEN_RES_REG;BEN_CMU_TOP;BEN_QLT_COD;BEN_SEX_COD;DDP_SPE_COD;ETE_CAT_SNDS;ETE_REG_COD;ETE_TYP_SNDS;ETP_REG_COD;ETP_CAT_SNDS;MDT_TYP_COD;MFT_COD;PRS_FJH_TYP;PRS_ACT_COG;PRS_ACT_NBR;PRS_ACT_QTE;PRS_DEP_MNT;PRS_PAI_MNT;PRS_REM_BSE;PRS_REM_MNT;FLT_ACT_COG;FLT_ACT_NBR;FLT_ACT_QTE;FLT_PAI_MNT;FLT_DEP_MNT;FLT_REM_MNT;SOI_ANN;SOI_MOI;ASU_NAT;ATT_NAT;CPL_COD;CPT_ENV_TYP;DRG_AFF_NAT;ETE_IND_TAA;EXO_MTF;MTM_NAT;PRS_NAT;PRS_PPU_SEC;PRS_REM_TAU;PRS_REM_TYP;PRS_PDS_QCP;EXE_INS_REG;PSE_ACT_SNDS;PSE_ACT_CAT;PSE_SPE_SNDS;PSE_STJ_SNDS;PRE_INS_REG;PSP_ACT_SNDS;PSP_ACT_CAT;PSP_SPE_SNDS;PSP_STJ_SNDS;TOP_PS5_TRG;
201612;76;20;76;9;1;1;0;1101;76;1;76;1101;9;3;0;2;1;1;0;46;46;46;2;1;1;46;0;46;2016;12;40;0;0;9;99;1;99;9;1117;1;100;99;3;99;0;0;14;2;99;0;0;1;2;9;
201612;28;30;28;9;1;2;0;9999;99;99;24;1102;9;99;0;1;5;1;0;.51;.51;.51;1;5;1;.51;0;.51;2016;12;10;0;0;9;99;9;99;9;3380;2;100;99;31;28;50;2;0;1;99;0;0;6;2;9;
On calcule le nombre de lignes :
with open(filename, "r") as f:
nb = 0
for line in f:
nb += 1
nb
199999
On peut aussi utiliser pandas.read_csv pour regarder les premières lignes.
import time
import pandas
begin = time.perf_counter()
df = pandas.read_csv(filename, sep=";", nrows=100)
print(time.perf_counter() - begin)
0.02099410000002422
df.head()
| FLX_ANN_MOI | ORG_CLE_REG | AGE_BEN_SNDS | BEN_RES_REG | BEN_CMU_TOP | BEN_QLT_COD | BEN_SEX_COD | DDP_SPE_COD | ETE_CAT_SNDS | ETE_REG_COD | ... | PSE_ACT_CAT | PSE_SPE_SNDS | PSE_STJ_SNDS | PRE_INS_REG | PSP_ACT_SNDS | PSP_ACT_CAT | PSP_SPE_SNDS | PSP_STJ_SNDS | TOP_PS5_TRG | Unnamed: 55 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 201612 | 76 | 20 | 76 | 9 | 1 | 1 | 0 | 1101 | 76 | ... | 0 | 14 | 2 | 99 | 0 | 0 | 1 | 2 | 9 | NaN |
| 1 | 201612 | 28 | 30 | 28 | 9 | 1 | 2 | 0 | 9999 | 99 | ... | 2 | 0 | 1 | 99 | 0 | 0 | 6 | 2 | 9 | NaN |
| 2 | 201612 | 27 | 60 | 93 | 9 | 2 | 1 | 0 | 9999 | 99 | ... | 2 | 0 | 9 | 93 | 0 | 1 | 1 | 9 | 9 | NaN |
| 3 | 201612 | 32 | 40 | 32 | 9 | 2 | 2 | 0 | 9999 | 99 | ... | 1 | 1 | 1 | 32 | 0 | 1 | 1 | 1 | 9 | NaN |
| 4 | 201612 | 32 | 30 | 32 | 9 | 1 | 2 | 0 | 2206 | 32 | ... | 0 | 0 | 2 | 99 | 0 | 0 | 1 | 2 | 9 | NaN |
5 rows × 56 columns
df.head().T
| 0 | 1 | 2 | 3 | 4 | |
|---|---|---|---|---|---|
| FLX_ANN_MOI | 201612.0 | 201612.00 | 201612.0 | 201612.0 | 201612.0 |
| ORG_CLE_REG | 76.0 | 28.00 | 27.0 | 32.0 | 32.0 |
| AGE_BEN_SNDS | 20.0 | 30.00 | 60.0 | 40.0 | 30.0 |
| BEN_RES_REG | 76.0 | 28.00 | 93.0 | 32.0 | 32.0 |
| BEN_CMU_TOP | 9.0 | 9.00 | 9.0 | 9.0 | 9.0 |
| BEN_QLT_COD | 1.0 | 1.00 | 2.0 | 2.0 | 1.0 |
| BEN_SEX_COD | 1.0 | 2.00 | 1.0 | 2.0 | 2.0 |
| DDP_SPE_COD | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| ETE_CAT_SNDS | 1101.0 | 9999.00 | 9999.0 | 9999.0 | 2206.0 |
| ETE_REG_COD | 76.0 | 99.00 | 99.0 | 99.0 | 32.0 |
| ETE_TYP_SNDS | 1.0 | 99.00 | 99.0 | 99.0 | 6.0 |
| ETP_REG_COD | 76.0 | 24.00 | 99.0 | 99.0 | 32.0 |
| ETP_CAT_SNDS | 1101.0 | 1102.00 | 9999.0 | 9999.0 | 2206.0 |
| MDT_TYP_COD | 9.0 | 9.00 | 9.0 | 9.0 | 9.0 |
| MFT_COD | 3.0 | 99.00 | 99.0 | 99.0 | 36.0 |
| PRS_FJH_TYP | 0.0 | 0.00 | 8.0 | 0.0 | 0.0 |
| PRS_ACT_COG | 2.0 | 1.00 | 1.0 | 2.0 | 1.0 |
| PRS_ACT_NBR | 1.0 | 5.00 | NaN | 2.0 | 1.0 |
| PRS_ACT_QTE | 1.0 | 1.00 | 1.0 | 2.0 | 1.0 |
| PRS_DEP_MNT | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| PRS_PAI_MNT | 46.0 | 0.51 | 0.0 | 46.0 | 23.0 |
| PRS_REM_BSE | 46.0 | 0.51 | 0.0 | 46.0 | 23.0 |
| PRS_REM_MNT | 46.0 | 0.51 | -0.5 | 32.2 | 23.0 |
| FLT_ACT_COG | 2.0 | 1.00 | 1.0 | 2.0 | 1.0 |
| FLT_ACT_NBR | 1.0 | 5.00 | NaN | 2.0 | 1.0 |
| FLT_ACT_QTE | 1.0 | 1.00 | 1.0 | 2.0 | 1.0 |
| FLT_PAI_MNT | 46.0 | 0.51 | 0.0 | 46.0 | 23.0 |
| FLT_DEP_MNT | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| FLT_REM_MNT | 46.0 | 0.51 | -0.5 | 32.2 | 23.0 |
| SOI_ANN | 2016.0 | 2016.00 | 2016.0 | 2016.0 | 2016.0 |
| SOI_MOI | 12.0 | 12.00 | 12.0 | 12.0 | 12.0 |
| ASU_NAT | 40.0 | 10.00 | 10.0 | 10.0 | 10.0 |
| ATT_NAT | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| CPL_COD | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| CPT_ENV_TYP | 9.0 | 9.00 | 9.0 | 9.0 | 9.0 |
| DRG_AFF_NAT | 99.0 | 99.00 | 99.0 | 99.0 | 99.0 |
| ETE_IND_TAA | 1.0 | 9.00 | 9.0 | 9.0 | 0.0 |
| EXO_MTF | 99.0 | 99.00 | 99.0 | 99.0 | 99.0 |
| MTM_NAT | 9.0 | 9.00 | 9.0 | 9.0 | 9.0 |
| PRS_NAT | 1117.0 | 3380.00 | 1972.0 | 1111.0 | 1111.0 |
| PRS_PPU_SEC | 1.0 | 2.00 | 2.0 | 2.0 | 2.0 |
| PRS_REM_TAU | 100.0 | 100.00 | 100.0 | 70.0 | 100.0 |
| PRS_REM_TYP | 99.0 | 99.00 | 99.0 | 99.0 | 99.0 |
| PRS_PDS_QCP | 3.0 | 31.00 | 31.0 | 10.0 | 31.0 |
| EXE_INS_REG | 99.0 | 28.00 | 93.0 | 32.0 | 99.0 |
| PSE_ACT_SNDS | 0.0 | 50.00 | 50.0 | 0.0 | 19.0 |
| PSE_ACT_CAT | 0.0 | 2.00 | 2.0 | 1.0 | 0.0 |
| PSE_SPE_SNDS | 14.0 | 0.00 | 0.0 | 1.0 | 0.0 |
| PSE_STJ_SNDS | 2.0 | 1.00 | 9.0 | 1.0 | 2.0 |
| PRE_INS_REG | 99.0 | 99.00 | 93.0 | 32.0 | 99.0 |
| PSP_ACT_SNDS | 0.0 | 0.00 | 0.0 | 0.0 | 0.0 |
| PSP_ACT_CAT | 0.0 | 0.00 | 1.0 | 1.0 | 0.0 |
| PSP_SPE_SNDS | 1.0 | 6.00 | 1.0 | 1.0 | 1.0 |
| PSP_STJ_SNDS | 2.0 | 2.00 | 9.0 | 1.0 | 2.0 |
| TOP_PS5_TRG | 9.0 | 9.00 | 9.0 | 9.0 | 9.0 |
| Unnamed: 55 | NaN | NaN | NaN | NaN | NaN |
variables = pandas.read_excel(xlsfile, skiprows=2)
cols = variables.columns[:2]
variables[cols].dropna()
| AGE_BEN_SNDS | Tranche d'Age Bénéficiaire au moment des soins | |
|---|---|---|
| 0 | ASU_NAT | Nature d'Assurance |
| 1 | ATT_NAT | Nature de l'Accident du Travail |
| 2 | BEN_CMU_TOP | Top Bénéficiaire CMU-C |
| 3 | BEN_QLT_COD | Qualité du Bénéficiaire |
| 4 | BEN_RES_ZEAT | ZEAT de Résidence du Bénéficiaire |
| 5 | BEN_SEX_COD | Sexe du Bénéficiaire |
| 6 | CPL_COD | Complément d'Acte |
| 7 | CPT_ENV_TYP | Type d'Enveloppe |
| 8 | DDP_SPE_COD | Discipline de Prestation Etb Exécutant |
| 9 | DRG_AFF_NAT | Nature du Destinataire de Règlement affiné |
| 10 | ETE_CAT_SNDS | Catégorie Etb Exécutant |
| 11 | ETE_IND_TAA | Indicateur TAA Privé/Public |
| 12 | ETE_TYP_SNDS | Type Etb Exécutant |
| 13 | ETE_ZEAT_COD | ZEAT d'Implantation Etb Exécutant |
| 14 | ETP_CAT_SNDS | Catégorie Etb Prescripteur |
| 15 | ETP_ZEAT_COD | ZEAT d'Implantation Etb Prescripteur |
| 16 | EXE_INS_ZEAT | ZEAT du PS Exécutant |
| 17 | EXO_MTF | Motif d'Exonération du Ticket Modérateur |
| 18 | FLT_ACT_COG | Coefficient Global de la Prestation Préfiltré |
| 19 | FLT_ACT_NBR | Dénombrement de la Prestation Préfiltré |
| 20 | FLT_ACT_QTE | Quantité de la Prestation Préfiltrée |
| 21 | FLT_DEP_MNT | Montant du Dépassement de la Prestation Préfiltré |
| 22 | FLT_PAI_MNT | Montant de la Dépense de la Prestation Préfiltrée |
| 23 | FLT_REM_MNT | Montant Versé/Remboursé Préfiltré |
| 24 | FLX_ANN_MOI | Année et Mois de Traitement |
| 25 | MDT_TYP_COD | Mode de Traitement Etb Exécutant |
| 26 | MFT_COD | Mode de Fixation des Tarifs Etb Exécutant |
| 27 | MTM_NAT | Modulation du Ticket Modérateur |
| 28 | ORG_CLE_ZEAT | ZEAT de l'Organisme de Liquidation |
| 29 | PRE_INS_ZEAT | ZEAT du PS Prescripteur |
| 30 | PRS_ACT_COG | Coefficient Global |
| 31 | PRS_ACT_NBR | Dénombrement |
| 32 | PRS_ACT_QTE | Quantité |
| 33 | PRS_DEP_MNT | Montant du Dépassement |
| 34 | PRS_FJH_TYP | Type de Prise en Charge Forfait Journalier |
| 35 | PRS_NAT | Nature de Prestation |
| 36 | PRS_PAI_MNT | Montant de la Dépense |
| 37 | PRS_PDS_QCP | Code Qualificatif Parcours de Soins (sortie) |
| 38 | PRS_PPU_SEC | Code Secteur Privé/Public |
| 39 | PRS_REM_BSE | Base de Remboursement |
| 40 | PRS_REM_MNT | Montant Versé/Remboursé |
| 41 | PRS_REM_TAU | Taux de Remboursement |
| 42 | PRS_REM_TYP | Type de Remboursement |
| 43 | PSE_ACT_CAT | Catégorie de l' Exécutant |
| 44 | PSE_ACT_SNDS | Nature d'Activité PS Exécutant |
| 45 | PSE_SPE_SNDS | Spécialité Médicale PS Exécutant |
| 46 | PSE_STJ_SNDS | Statut Juridique PS Exécutant |
| 47 | PSP_ACT_CAT | Catégorie du Prescripteur |
| 48 | PSP_ACT_SNDS | Nature d'Activité PS Prescripteur |
| 49 | PSP_SPE_SNDS | Spécialité Médicale PS Prescripteur |
| 50 | PSP_STJ_SNDS | Statut Juridique PS Prescripteur |
| 51 | SOI_ANN | Année de Soins |
| 52 | SOI_MOI | Mois de Soins |
| 53 | TOP_PS5_TRG | Top Périmètre hors CMU C et prestations pour i... |
Répartition des dépenses selon l’âge#
C’est la variable AGE_BEN_SNDS mais on ne peut toujours pas charger
le fichier en mémoire.
Itérateur sur des dataframes#
Il faut utiliser le paramètre iterator.
dfs = []
for i, df in enumerate(pandas.read_csv(filename,
sep=";", iterator=True, chunksize=10000)):
dfs.append(df)
if i >= 5:
break
dfs[0].head()
| FLX_ANN_MOI | ORG_CLE_REG | AGE_BEN_SNDS | BEN_RES_REG | BEN_CMU_TOP | BEN_QLT_COD | BEN_SEX_COD | DDP_SPE_COD | ETE_CAT_SNDS | ETE_REG_COD | ... | PSE_ACT_CAT | PSE_SPE_SNDS | PSE_STJ_SNDS | PRE_INS_REG | PSP_ACT_SNDS | PSP_ACT_CAT | PSP_SPE_SNDS | PSP_STJ_SNDS | TOP_PS5_TRG | Unnamed: 55 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 201612 | 76 | 20 | 76 | 9 | 1 | 1 | 0 | 1101 | 76 | ... | 0 | 14 | 2 | 99 | 0 | 0 | 1 | 2 | 9 | NaN |
| 1 | 201612 | 28 | 30 | 28 | 9 | 1 | 2 | 0 | 9999 | 99 | ... | 2 | 0 | 1 | 99 | 0 | 0 | 6 | 2 | 9 | NaN |
| 2 | 201612 | 27 | 60 | 93 | 9 | 2 | 1 | 0 | 9999 | 99 | ... | 2 | 0 | 9 | 93 | 0 | 1 | 1 | 9 | 9 | NaN |
| 3 | 201612 | 32 | 40 | 32 | 9 | 2 | 2 | 0 | 9999 | 99 | ... | 1 | 1 | 1 | 32 | 0 | 1 | 1 | 1 | 9 | NaN |
| 4 | 201612 | 32 | 30 | 32 | 9 | 1 | 2 | 0 | 2206 | 32 | ... | 0 | 0 | 2 | 99 | 0 | 0 | 1 | 2 | 9 | NaN |
5 rows × 56 columns
Il faut calculer la distribution des âges pour chaque dataframe.
from datetime import datetime
ages = []
for i, df in enumerate(pandas.read_csv(filename,
sep=";", iterator=True, chunksize=10000)):
sub = df[["AGE_BEN_SNDS", "PRS_REM_MNT"]].copy()
gr = sub.groupby("AGE_BEN_SNDS").sum()
ages.append(gr)
if len(ages) % 100 == 0:
print(datetime.now(), len(ages), "lignes", 10000 * len(ages))
Les versions de pandas ont quelques différences. Voici celle utilisées pour ce notebook.
pandas.__version__
'1.1.0'
age = pandas.concat(ages)
agg = age.groupby("AGE_BEN_SNDS").sum()
agg.plot(kind="bar");
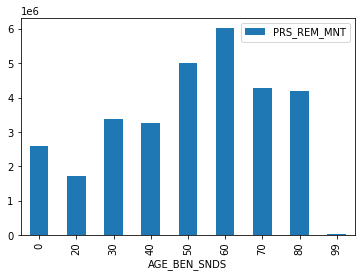
Difficile d’interpréter ce graphique sans le comparer à la pyramide des âges mais ce n’est pas l’objectif ici. Chaque statistique prend un peu de temps. Et si nous tirions un échantillon aléatoire à 1%.
Echantillon#
ech = []
for i, df in enumerate(pandas.read_csv(filename,
sep=";", iterator=True, chunksize=10000)):
sub = df.sample(frac=0.01)
ech.append(sub)
if len(ech) % 100 == 0:
print(datetime.now(), len(ech), "lignes", 10000 * len(ech))
echantillon = pandas.concat(ech)
echantillon.shape
(1000, 56)
echantillon[["AGE_BEN_SNDS", "PRS_REM_MNT"]] \
.groupby("AGE_BEN_SNDS").sum().plot(kind="bar");
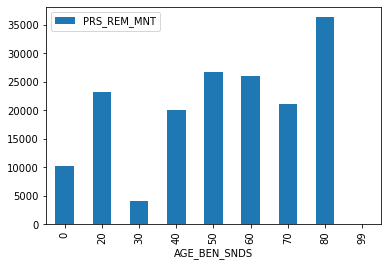
pickle#
Le module pickle permet de sérialiser. Cette opération transforme le contenu d’un objet tel qu’il est en mémoire en une séquence d’octets qu’on peut alors enregistrer sur disque. Comme ce format de fichier est plus proche de la mémoire, il se charge beaucoup plus vite. Cela permet de gagner un peu de temps. L’inconvénient est que ce format n’est pas toujours compatible d’une version de python ou pandas à l’autre.
echantillon.to_csv("echantillon.csv", sep=";", index=False)
import os
os.stat("echantillon.csv").st_size / 2**20
0.16968822479248047
begin = time.perf_counter()
ech2 = pandas.read_csv("echantillon.csv", sep=";")
print(time.perf_counter()-begin)
0.025167299999992565
ech2.shape
(1000, 56)
echantillon.to_pickle("echantillon.pickle")
begin = time.perf_counter()
ech3 = pandas.read_pickle("echantillon.pickle")
print(time.perf_counter()-begin)
0.011920500000002221
SQLite3#
Un échantillon fait disparaître les classes les moins représentées. On peut accélérer le traitement en passant par Sqlite et disponible via le module sqlite. Première étape : créer la base.
import sqlite3
cnx = sqlite3.connect('damir2.db3')
for i, df in enumerate(pandas.read_csv(filename,
sep=";", iterator=True, chunksize=100000)):
df.to_sql(con=cnx, name="damir", if_exists="replace" if i == 0 else "append")
if (i+1) % 10 == 0:
print(datetime.now(), (i+1) * 100000, "lignes")
print("done")
c:python372_x64libsite-packagespandascoregeneric.py:2612: UserWarning: The spaces in these column names will not be changed. In pandas versions < 0.14, spaces were converted to underscores. method=method,
done
C’est un peu long mais le calcul de statistique est plus rapide par la suite.
import time
begin = time.perf_counter()
dfage = pandas.read_sql(
'select AGE_BEN_SNDS, count(*) from damir group by AGE_BEN_SNDS', cnx)
print("time", time.perf_counter() - begin)
dfage
time 0.08782719999999244
| AGE_BEN_SNDS | count(*) | |
|---|---|---|
| 0 | 0 | 10424 |
| 1 | 20 | 10948 |
| 2 | 30 | 10871 |
| 3 | 40 | 11963 |
| 4 | 50 | 14475 |
| 5 | 60 | 15662 |
| 6 | 70 | 13220 |
| 7 | 80 | 12281 |
| 8 | 99 | 155 |
On peut aussi programmer des commandes magiques pour alléger la syntaxe.
%load_ext pyensae
%SQL_connect damir.db3
<pyensae.sql.sql_interface_database.InterfaceSQLDatabase at 0x1dc5c25add8>
%SQL_tables
[]
%%SQL
SELECT * FROM damir LIMIT 5
unable to execute a SQL request (1)(file damir.db3)
SELECT * FROM damir LIMIT 5
no such table: damir
'SELECT * FROM damir LIMIT 5'
%lsmagic
Available line magics:
%SQL %SQL_add_function %SQL_close %SQL_connect %SQL_drop_table %SQL_import_df %SQL_import_tsv %SQL_refresh_completion %SQL_schema %SQL_tables %alias %alias_magic %autoawait %autocall %automagic %autosave %bookmark %cd %clear %cls %colors %conda %config %connect_info %copy %ddir %debug %dhist %dirs %doctest_mode %echo %ed %edit %encoding %env %grep %gui %head %hhelp %hist %history %jsdf %killbgscripts %ldir %less %load %load_ext %loadpy %logoff %logon %logstart %logstate %logstop %ls %lsmagic %lsr %lsrepo %macro %magic %matplotlib %mkdir %more %mpl_style %nb_menu %notebook %page %pastebin %pdb %pdef %pdoc %pfile %pinfo %pinfo2 %pip %popd %pprint %precision %prun %psearch %psource %pushd %pwd %pycat %pylab %qtconsole %quickref %recall %rehashx %reload_ext %ren %rep %rerun %reset %reset_selective %rmdir %run %save %sc %set_env %store %sx %system %tail %tb %time %timeit %unalias %unload_ext %who %who_ls %whos %xdel %xmode
Available cell magics:
%%! %%HTML %%PYTHON %%SQL %%SVG %%bash %%capture %%cmd %%debug %%file %%html %%javascript %%js %%latex %%markdown %%perl %%prun %%pypy %%python %%python2 %%python3 %%ruby %%runpy %%script %%sh %%svg %%sx %%system %%time %%timeit %%writefile
Automagic is ON, % prefix IS NOT needed for line magics.
dask#
dask fonctionne un peu comme pandas excepté que l’exécution des requêtes est différée. On utilise read_csv.
import dask.dataframe
dfdk = dask.dataframe.read_csv(filename, sep=";")
dfdk.head(n=2)
| FLX_ANN_MOI | ORG_CLE_REG | AGE_BEN_SNDS | BEN_RES_REG | BEN_CMU_TOP | BEN_QLT_COD | BEN_SEX_COD | DDP_SPE_COD | ETE_CAT_SNDS | ETE_REG_COD | ... | PSE_ACT_CAT | PSE_SPE_SNDS | PSE_STJ_SNDS | PRE_INS_REG | PSP_ACT_SNDS | PSP_ACT_CAT | PSP_SPE_SNDS | PSP_STJ_SNDS | TOP_PS5_TRG | Unnamed: 55 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 201612 | 76 | 20 | 76 | 9 | 1 | 1 | 0 | 1101 | 76 | ... | 0 | 14 | 2 | 99 | 0 | 0 | 1 | 2 | 9 | NaN |
| 1 | 201612 | 28 | 30 | 28 | 9 | 1 | 2 | 0 | 9999 | 99 | ... | 2 | 0 | 1 | 99 | 0 | 0 | 6 | 2 | 9 | NaN |
2 rows × 56 columns
Les trois lignes qui suivent sont très rapides.
import dask
dask.__version__
'2.12.0'
dfdk2 = dfdk[["AGE_BEN_SNDS", "PRS_REM_MNT"]]
grdk = dfdk2.groupby("AGE_BEN_SNDS").sum()
La suivante, beaucoup moins.
begin = time.perf_counter()
res = grdk.compute()
print(time.perf_counter() - begin)
0.5088113999999848
ech4 = dfdk.sample(frac=0.01)
dfech = ech4.compute()
type(dfech)
pandas.core.frame.DataFrame
res
| PRS_REM_MNT | |
|---|---|
| AGE_BEN_SNDS | |
| 0 | 2607345.16 |
| 20 | 1714894.46 |
| 30 | 3378001.46 |
| 40 | 3271108.13 |
| 50 | 5011475.59 |
| 60 | 6026792.72 |
| 70 | 4283277.72 |
| 80 | 4181565.36 |
| 99 | 19015.33 |
C’est plus qu’avec SQLite3 mais dans cet exemple, dask relit le fichier à chaque calcul car il ne stocke pas un dataframe dans un format intermédiaire plus efficace tel que parquet. dask propose des idées pour optimiser les calculs Dask DataFrame Performance Tips.
Grandes matrices#
Les dataframes acceptent un type différent pour chaque colonne. Si toutes les colonnes sont numériques et de même types, on peut s’orienter vers d’autres solutions.
Zarr#
zarr permet de manipuler des matrices qui ne tiennent pas en mémoire de la même façon que numpy. Les données sont compressés et parfois stockées sur disque. Ce module masque beaucoup de détails en proposant une interface connue.
import zarr
z = zarr.zeros((10000, 10000), chunks=(1000, 1000), dtype='i4')
z
<zarr.core.Array (10000, 10000) int32>
z[4,5]
0
Voir aussi wendelin.core, hdf5.