Tech - manipulation de données avec pandas#
Links: notebook, html, python, slides, GitHub
pandas est la librairie incontournable pour manipuler les données. Elle permet de manipuler aussi bien les données sous forme de tables qu’elle peut récupérer ou exporter en différents formats. Elle permet également de créer facilement des graphes.
from jyquickhelper import add_notebook_menu
add_notebook_menu()
%matplotlib inline
Enoncé#
La librairie pandas implémente la classe DataFrame. C’est une structure de table, chaque colonne porte un nom et contient un seul type de données. C’est très similaire au langage SQL.
Création d’un dataframe#
Il existe une grande variété pour créer un DataFrame. Voici les deux principaux. Le premier : une liste de dictionnaires. Chaque clé est le nom de la colonne.
from pandas import DataFrame
rows = [{'col1': 0.5, 'col2': 'schtroumph'},
{'col1': 0.6, 'col2': 'schtroumphette'}]
DataFrame(rows)
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | schtroumph |
| 1 | 0.6 | schtroumphette |
La lecture depuis un fichier :
%%writefile data.csv
col1,col2
0.5,alpha
0.6,beta
Overwriting data.csv
import os
os.getcwd()
'C:\xavierdupre\__home_\GitHub\ensae_teaching_cs\_doc\notebooks\td1a_home'
from pandas import read_csv
df = read_csv('data.csv')
df
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
La maîtrise des index#
Les index fonctionnent à peu près comme numpy mais offre plus d’options puisque les colonnes mais aussi les lignes ont un nom.
Accès par colonne
df
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
df['col1']
0 0.5
1 0.6
Name: col1, dtype: float64
df[['col1', 'col2']]
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
Accès par ligne (uniquement avec :). On se sert principalement de
l’opérateur : pour les lignes.
df[:1]
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | alpha |
Accès par positions avec loc.
df.loc[0, 'col1']
0.5
Accès par positions entières avec iloc.
df.iloc[0, 0]
0.5
La maîtrise des index des lignes#
La création d’un dataframe donne l’impression que les index des lignes sont des entiers mais cela peut être changer
df
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
dfi = df.set_index('col2')
dfi
| col1 | |
|---|---|
| col2 | |
| alpha | 0.5 |
| beta | 0.6 |
dfi.loc['alpha', 'col1']
0.5
Il faut se souvenir de cette particularité lors de la fusion de tables.
La maîtrise des index des colonnes#
Les colonnes sont nommées.
df.columns
Index(['col1', 'col2'], dtype='object')
On peut les renommer.
df.columns = ["valeur", "nom"]
df
| valeur | nom | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
L’opérateur : peut également servir pour les colonnes.
df.loc[:, 'valeur':'nom']
| valeur | nom | |
|---|---|---|
| 0 | 0.5 | alpha |
| 1 | 0.6 | beta |
Lien vers numpy#
pandas utilise numpy pour stocker les données. Il est possible de récupérer des matrices depuis des DataFrame avec values.
df.values
array([[0.5, 'alpha'],
[0.6, 'beta']], dtype=object)
df[['valeur']].values
array([[0.5],
[0.6]])
La maîtrise du nan#
nan est une convention pour désigner une valeur manquante.
rows = [{'col1': 0.5, 'col2': 'schtroumph'},
{'col2': 'schtroumphette'}]
DataFrame(rows)
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | schtroumph |
| 1 | NaN | schtroumphette |
La maîtrise des types#
Un dataframe est défini par ses dimensions et chaque colonne a un type potentiellement différent.
df.dtypes
valeur float64
nom object
dtype: object
On peut changer un type, donc convertir toutes les valeurs d’une colonne vers un autre type.
import numpy
df['valeur'].astype(numpy.float32)
0 0.5
1 0.6
Name: valeur, dtype: float32
import numpy
df['valeur'].astype(numpy.int32)
0 0
1 0
Name: valeur, dtype: int32
Création de colonnes#
On peut facilement créer de nouvelles colonnes.
df['sup055'] = df['valeur'] >= 0.55
df
| valeur | nom | sup055 | |
|---|---|---|---|
| 0 | 0.5 | alpha | False |
| 1 | 0.6 | beta | True |
df['sup055'] = (df['valeur'] >= 0.55).astype(numpy.int64)
df
| valeur | nom | sup055 | |
|---|---|---|---|
| 0 | 0.5 | alpha | 0 |
| 1 | 0.6 | beta | 1 |
df['sup055+'] = df['valeur'] + df['sup055']
df
| valeur | nom | sup055 | sup055+ | |
|---|---|---|---|---|
| 0 | 0.5 | alpha | 0 | 0.5 |
| 1 | 0.6 | beta | 1 | 1.6 |
Modifications de valeurs#
On peut les modifier une à une en utilisant les index. Les notations sont souvent intuitives. Elles ne seront pas toutes détaillées. Ci-dessous un moyen de modifer certaines valeurs selon une condition.
df.loc[df['nom'] == 'alpha', 'sup055+'] += 1000
df
| valeur | nom | sup055 | sup055+ | |
|---|---|---|---|---|
| 0 | 0.5 | alpha | 0 | 1000.5 |
| 1 | 0.6 | beta | 1 | 1.6 |
Une erreur ou warning fréquent#
rows = [{'col1': 0.5, 'col2': 'schtroumph'},
{'col1': 1.5, 'col2': 'schtroumphette'}]
df = DataFrame(rows)
df
| col1 | col2 | |
|---|---|---|
| 0 | 0.5 | schtroumph |
| 1 | 1.5 | schtroumphette |
df1 = df[df['col1'] > 1.]
df1
| col1 | col2 | |
|---|---|---|
| 1 | 1.5 | schtroumphette |
df1["col3"] = df1["col1"] + 1.
df1
<ipython-input-31-e5d64c4890f8>:1: SettingWithCopyWarning:
A value is trying to be set on a copy of a slice from a DataFrame.
Try using .loc[row_indexer,col_indexer] = value instead
See the caveats in the documentation: https://pandas.pydata.org/pandas-docs/stable/user_guide/indexing.html#returning-a-view-versus-a-copy
df1["col3"] = df1["col1"] + 1.
| col1 | col2 | col3 | |
|---|---|---|---|
| 1 | 1.5 | schtroumphette | 2.5 |
A value is trying to be set on a copy of a slice from a DataFrame. :
Par défaut, l’instruction df[df['col1'] > 1.] ne crée pas un nouveau
DataFrame, elle crée ce qu’on appelle une vue pour éviter de copier les
données. Le résultat ne contient que l’index des lignes qui ont été
sélectionnées et un lien vers le dataframe original. L’avertissement
stipule que pandas ne peut pas modifier le dataframe original mais
qu’il doit effectuer une copie.
La solution pour faire disparaître ce warning est de copier le dataframe.
df2 = df1.copy()
df2["col3"] = df2["col1"] + 1.
La maîtrise des fonctions#
Les fonctions de pandas créent par défaut un nouveau dataframe plutôt que de modifier un dataframe existant. Cela explique pourquoi parfois la mémoire se retrouve congestionnée. La page 10 minutes to pandas est un bon début.
On récupère les données du COVID par région et par âge et premier graphe#
A cette adresse : Données hospitalières relatives à l’épidémie de COVID-19
# https://www.data.gouv.fr/en/datasets/r/63352e38-d353-4b54-bfd1-f1b3ee1cabd7
from pandas import read_csv
url = "https://www.data.gouv.fr/en/datasets/r/08c18e08-6780-452d-9b8c-ae244ad529b3"
covid = read_csv(url, sep=";")
covid.tail()
| reg | cl_age90 | jour | hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|---|---|---|
| 108697 | 94 | 59 | 2021-09-17 | 6 | 0 | 3.0 | 3.0 | 0.0 | 183 | 8 |
| 108698 | 94 | 69 | 2021-09-17 | 11 | 3 | 4.0 | 2.0 | 2.0 | 219 | 23 |
| 108699 | 94 | 79 | 2021-09-17 | 8 | 1 | 5.0 | 2.0 | 0.0 | 245 | 56 |
| 108700 | 94 | 89 | 2021-09-17 | 11 | 1 | 8.0 | 2.0 | 0.0 | 220 | 91 |
| 108701 | 94 | 90 | 2021-09-17 | 7 | 0 | 4.0 | 3.0 | 0.0 | 88 | 50 |
covid.dtypes
reg int64
cl_age90 int64
jour object
hosp int64
rea int64
HospConv float64
SSR_USLD float64
autres float64
rad int64
dc int64
dtype: object
Les dates sont considérées comme des chaînes de caractères. Il est plus simple pour réaliser des opérations de convertir la colonne sous forme de dates.
from pandas import to_datetime
covid['jour'] = to_datetime(covid['jour'])
covid.tail()
| reg | cl_age90 | jour | hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|---|---|---|
| 108697 | 94 | 59 | 2021-09-17 | 6 | 0 | 3.0 | 3.0 | 0.0 | 183 | 8 |
| 108698 | 94 | 69 | 2021-09-17 | 11 | 3 | 4.0 | 2.0 | 2.0 | 219 | 23 |
| 108699 | 94 | 79 | 2021-09-17 | 8 | 1 | 5.0 | 2.0 | 0.0 | 245 | 56 |
| 108700 | 94 | 89 | 2021-09-17 | 11 | 1 | 8.0 | 2.0 | 0.0 | 220 | 91 |
| 108701 | 94 | 90 | 2021-09-17 | 7 | 0 | 4.0 | 3.0 | 0.0 | 88 | 50 |
covid.dtypes
reg int64
cl_age90 int64
jour datetime64[ns]
hosp int64
rea int64
HospConv float64
SSR_USLD float64
autres float64
rad int64
dc int64
dtype: object
On supprime les colonnes relatives aux régions et à l’âge puis on aggrège par jour.
agg_par_jour = covid.drop(['reg', 'cl_age90'], axis=1).groupby('jour').sum()
agg_par_jour.tail()
| hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|
| jour | |||||||
| 2021-09-13 | 19929 | 4202 | 9829.0 | 5408.0 | 490.0 | 825603 | 177260 |
| 2021-09-14 | 19433 | 3996 | 9552.0 | 5383.0 | 502.0 | 827024 | 177450 |
| 2021-09-15 | 19065 | 3914 | 9264.0 | 5385.0 | 502.0 | 828103 | 177604 |
| 2021-09-16 | 18550 | 3900 | 8769.0 | 5379.0 | 502.0 | 829146 | 177733 |
| 2021-09-17 | 18096 | 3777 | 8472.0 | 5347.0 | 500.0 | 830154 | 177866 |
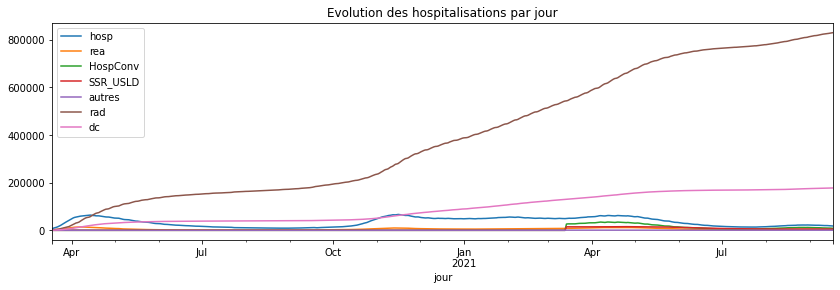
agg_par_jour.plot(title="Evolution des hospitalisations par jour",
figsize=(14, 4));
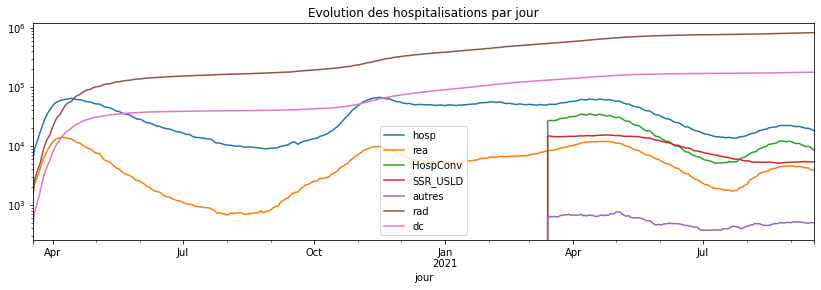
Avec échelle logarithmique.
agg_par_jour.plot(title="Evolution des hospitalisations par jour",
figsize=(14, 4), logy=True);
Q1 : refaire le graphique précédent pour votre classe d’âge#
Q2 : faire de même avec les séries différenciées#
Q3 : faire de même avec des séries lissées sur sur 7 jours#
Q4 : fusion de tables par départements#
Réponses#
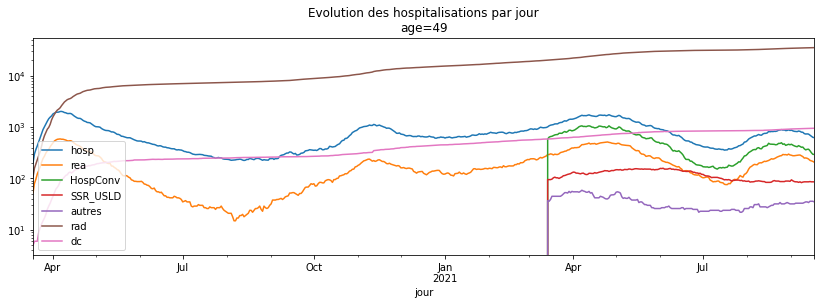
Q1 : refaire le graphique précédent pour votre classe d’âge#
set(covid['cl_age90'])
{0, 9, 19, 29, 39, 49, 59, 69, 79, 89, 90}
covid49 = covid[covid.cl_age90 == 49]
agg_par_jour49 = covid49.drop(['reg', 'cl_age90'], axis=1).groupby('jour').sum()
agg_par_jour49.tail()
| hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|
| jour | |||||||
| 2021-09-13 | 730 | 239 | 371.0 | 86.0 | 34.0 | 34875 | 942 |
| 2021-09-14 | 695 | 225 | 347.0 | 87.0 | 36.0 | 34969 | 945 |
| 2021-09-15 | 659 | 220 | 317.0 | 86.0 | 36.0 | 35044 | 946 |
| 2021-09-16 | 632 | 215 | 295.0 | 86.0 | 36.0 | 35096 | 947 |
| 2021-09-17 | 622 | 207 | 294.0 | 86.0 | 35.0 | 35144 | 949 |
agg_par_jour49.plot(title="Evolution des hospitalisations par jour\nage=49",
figsize=(14, 4), logy=True);
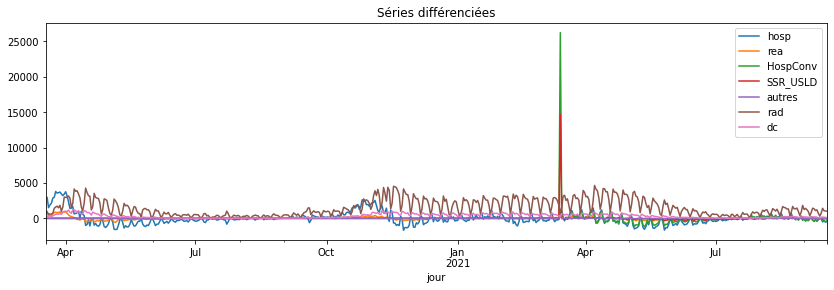
Q2 : faire de même avec les séries différenciées#
covid.tail()
| reg | cl_age90 | jour | hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|---|---|---|
| 108697 | 94 | 59 | 2021-09-17 | 6 | 0 | 3.0 | 3.0 | 0.0 | 183 | 8 |
| 108698 | 94 | 69 | 2021-09-17 | 11 | 3 | 4.0 | 2.0 | 2.0 | 219 | 23 |
| 108699 | 94 | 79 | 2021-09-17 | 8 | 1 | 5.0 | 2.0 | 0.0 | 245 | 56 |
| 108700 | 94 | 89 | 2021-09-17 | 11 | 1 | 8.0 | 2.0 | 0.0 | 220 | 91 |
| 108701 | 94 | 90 | 2021-09-17 | 7 | 0 | 4.0 | 3.0 | 0.0 | 88 | 50 |
diff = covid.drop(['reg', 'cl_age90'], axis=1).groupby(
['jour']).sum().diff()
diff.tail(n=2)
| hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|
| jour | |||||||
| 2021-09-16 | -515.0 | -14.0 | -495.0 | -6.0 | 0.0 | 1043.0 | 129.0 |
| 2021-09-17 | -454.0 | -123.0 | -297.0 | -32.0 | -2.0 | 1008.0 | 133.0 |
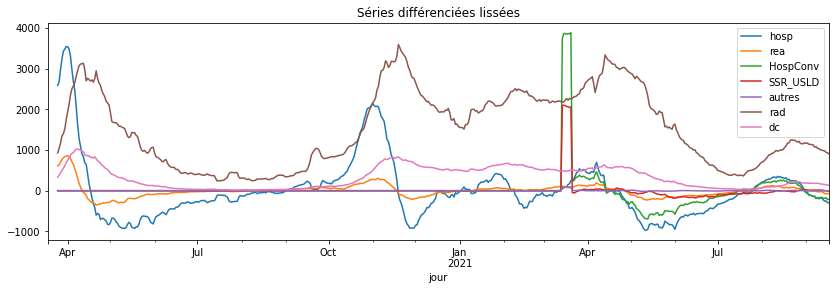
diff.plot(title="Séries différenciées", figsize=(14, 4));
Q3 : faire de même avec des séries lissées sur sur 7 jours#
diff.rolling(7)
Rolling [window=7,center=False,axis=0,method=single]
roll = diff.rolling(7).mean()
roll.tail(n=2)
| hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|
| jour | |||||||
| 2021-09-16 | -292.428571 | -68.857143 | -217.285714 | -5.428571 | -0.857143 | 933.571429 | 135.857143 |
| 2021-09-17 | -298.000000 | -74.000000 | -209.571429 | -13.857143 | -0.571429 | 904.714286 | 132.285714 |
roll.plot(title="Séries différenciées lissées", figsize=(14, 4));
Petit aparté#
On veut savoir combien de temps les gens restent à l’hôpital avant de sortir, en supposant que le temps de guérison est à peu près identique au temps passé lorsque l’issue est tout autre. Je pensais calculer les corrélations entre la série des décès et celles de réanimations décalées de plusieurs jours en me disant qu’un pic de corrélation pourrait indiquer une sorte de durée moyenne de réanimation.
data = agg_par_jour49.diff().rolling(7).mean()
data.tail(n=2)
| hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|
| jour | |||||||
| 2021-09-16 | -22.857143 | -7.285714 | -16.142857 | 0.142857 | 0.428571 | 59.714286 | 1.285714 |
| 2021-09-17 | -17.857143 | -6.000000 | -12.000000 | 0.000000 | 0.142857 | 54.571429 | 1.571429 |
data_last = data.tail(n=90)
cor = []
for i in range(0, 35):
ts = DataFrame(dict(rea=data_last.rea, dc=data_last.dc,
dclag=data_last["dc"].shift(i),
realag=data_last["rea"].shift(i)))
ts_cor = ts.corr()
cor.append(dict(delay=i, corr_dc=ts_cor.iloc[1, 3],
corr_rea=ts_cor.iloc[0, 3]))
DataFrame(cor).set_index('delay').plot(title="Corrélation entre décès et réanimation");
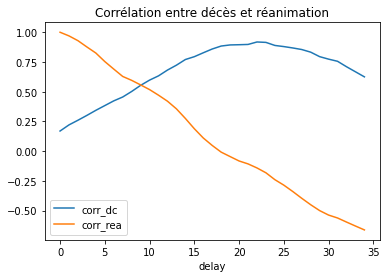
Il apparaît que ces corrélations sont très différentes selon qu’on les calcule sur les dernières données et les premières semaines. Cela semblerait indiquer que les données médicales sont très différentes. On pourrait chercher plusieurs jours mais le plus simple serait sans de générer des données artificielles avec un modèle SIR et vérifier si ce raisonnement tient la route sur des données propres.
Q4 : fusion de tables par départements#
On récupère deux jeux de données : * Données hospitalières relatives à l’épidémie de COVID-19 * Indicateurs de suivi de l’épidémie de COVID-19
hosp = read_csv("https://www.data.gouv.fr/en/datasets/r/63352e38-d353-4b54-bfd1-f1b3ee1cabd7",
sep=";")
hosp.tail()
| dep | sexe | jour | hosp | rea | HospConv | SSR_USLD | autres | rad | dc | |
|---|---|---|---|---|---|---|---|---|---|---|
| 167440 | 976 | 0 | 2021-09-17 | 7 | 1 | 6.0 | 0.0 | 0.0 | 1288 | 133 |
| 167441 | 976 | 1 | 2021-09-17 | 4 | 0 | 4.0 | 0.0 | 0.0 | 583 | 78 |
| 167442 | 976 | 2 | 2021-09-17 | 3 | 1 | 2.0 | 0.0 | 0.0 | 684 | 54 |
| 167443 | 978 | 0 | 2021-09-17 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0 | 1 |
| 167444 | 978 | 1 | 2021-09-17 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0 | 1 |
indic = read_csv("https://www.data.gouv.fr/fr/datasets/r/4acad602-d8b1-4516-bc71-7d5574d5f33e",
encoding="ISO-8859-1")
indic.tail()
| extract_date | departement | region | libelle_reg | libelle_dep | tx_incid | R | taux_occupation_sae | tx_pos | tx_incid_couleur | R_couleur | taux_occupation_sae_couleur | tx_pos_couleur | nb_orange | nb_rouge | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 55444 | 2020-11-08 | 84 | 93 | Provence Alpes Côte d'Azur | Vaucluse | 641.89 | NaN | 107.2 | 17.565854 | rouge | NaN | rouge | rouge | 0 | 3 |
| 55445 | 2020-10-26 | 84 | 93 | Provence Alpes Côte d'Azur | Vaucluse | 540.47 | NaN | 65.9 | 18.461913 | rouge | NaN | rouge | rouge | 0 | 3 |
| 55446 | 2020-10-27 | 84 | 93 | Provence Alpes Côte d'Azur | Vaucluse | 626.21 | 1.48 | 66.5 | 19.402408 | rouge | orange | rouge | rouge | 1 | 3 |
| 55447 | 2020-10-28 | 84 | 93 | Provence Alpes Côte d'Azur | Vaucluse | 672.20 | NaN | 68.5 | 19.795276 | rouge | NaN | rouge | rouge | 0 | 3 |
| 55448 | 2020-10-25 | 84 | 93 | Provence Alpes Côte d'Azur | Vaucluse | 439.75 | NaN | 61.3 | 16.519352 | rouge | NaN | rouge | rouge | 0 | 3 |
Le code suivant explique comment trouver la valeur ISO-8859-1.
# import chardet
# with open("indicateurs-covid19-dep.csv", "rb") as f:
# content = f.read()
# chardet.detect(content) # {'encoding': 'ISO-8859-1', 'confidence': 0.73, 'language': ''}
Q5 : une carte ?#
Tracer une carte n’est jamais simple. Il faut tout d’abord récupérer les coordonnées des départements : Contours des départements français issus d’OpenStreetMap. Ensuite… de ces fichiers ont été extraits les barycentres de chaque département français : departement_french_2018.csv. Ce fichier a été créé avec la fonction implémentée dans le fichier data_shape_files.py. Ce qui suit est une approximation de carte : on suppose que là où se trouve, les coordonnées longitude et latitude ne sont pas trop éloignées de ce qu’elles pourraient être si elles étaient projetées sur une sphère.
dep_pos = read_csv("https://raw.githubusercontent.com/sdpython/ensae_teaching_cs/"
"master/src/ensae_teaching_cs/data/data_shp/departement_french_2018.csv")
dep_pos.tail()
| code_insee | nom | nuts3 | wikipedia | surf_km2 | DEPLONG | DEPLAT | |
|---|---|---|---|---|---|---|---|
| 97 | 56 | Morbihan | FR524 | fr:Morbihan | 6870.0 | -2.812320 | 47.846846 |
| 98 | 25 | Doubs | FR431 | fr:Doubs (département) | 5256.0 | 6.362722 | 47.165964 |
| 99 | 39 | Jura | FR432 | fr:Jura (département) | 5049.0 | 5.697361 | 46.729368 |
| 100 | 07 | Ardèche | FR712 | fr:Ardèche (département) | 5566.0 | 4.425582 | 44.752771 |
| 101 | 30 | Gard | FR812 | fr:Gard | 5875.0 | 4.179861 | 43.993601 |
last_extract_date = max(set(indic.extract_date))
last_extract_date
'2021-09-17'
indic_last = indic[indic.extract_date == last_extract_date]
merge = indic_last.merge(dep_pos, left_on='departement', right_on='code_insee')
final = merge[['code_insee', 'nom', 'DEPLONG', 'DEPLAT', 'taux_occupation_sae', 'R']]
metro = final[final.DEPLAT > 40]
metro
| code_insee | nom | DEPLONG | DEPLAT | taux_occupation_sae | R | |
|---|---|---|---|---|---|---|
| 0 | 01 | Ain | 5.348764 | 46.099799 | 30.2 | NaN |
| 1 | 03 | Allier | 3.187644 | 46.393637 | 30.2 | NaN |
| 2 | 07 | Ardèche | 4.425582 | 44.752771 | 30.2 | NaN |
| 3 | 15 | Cantal | 2.669045 | 45.051247 | 30.2 | NaN |
| 4 | 26 | Drôme | 5.167364 | 44.685239 | 30.2 | NaN |
| ... | ... | ... | ... | ... | ... | ... |
| 95 | 05 | Hautes-Alpes | 6.265318 | 44.663965 | 69.1 | NaN |
| 96 | 06 | Alpes-Maritimes | 7.116532 | 43.937937 | 69.1 | NaN |
| 97 | 13 | Bouches-du-Rhône | 5.086225 | 43.543055 | 69.1 | NaN |
| 98 | 83 | Var | 6.244490 | 43.441656 | 69.1 | NaN |
| 99 | 84 | Vaucluse | 5.177329 | 44.007176 | 69.1 | NaN |
95 rows × 6 columns
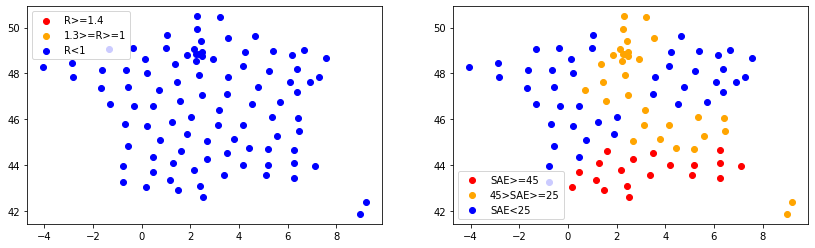
import matplotlib.pyplot as plt
fig, ax = plt.subplots(1, 2, figsize=(14, 4))
bigR1 = metro.R >= 1
bigR2 = metro.R >= 1.4
ax[0].scatter(metro.loc[bigR2, 'DEPLONG'], metro.loc[bigR2, 'DEPLAT'], c='red', label='R>=1.4');
ax[0].scatter(metro.loc[bigR1 & ~bigR2, 'DEPLONG'], metro.loc[bigR1 & ~bigR2, 'DEPLAT'], c='orange', label='1.3>=R>=1');
ax[0].scatter(metro.loc[~bigR1, 'DEPLONG'], metro.loc[~bigR1, 'DEPLAT'], c='blue', label='R<1');
ax[0].legend()
bigR1 = metro.taux_occupation_sae >= 25
bigR2 = metro.taux_occupation_sae >= 45
ax[1].scatter(metro.loc[bigR2, 'DEPLONG'], metro.loc[bigR2, 'DEPLAT'], c='red', label='SAE>=45');
ax[1].scatter(metro.loc[bigR1 & ~bigR2, 'DEPLONG'], metro.loc[bigR1 & ~bigR2, 'DEPLAT'], c='orange', label='45>SAE>=25');
ax[1].scatter(metro.loc[~bigR1, 'DEPLONG'], metro.loc[~bigR1, 'DEPLAT'], c='blue', label='SAE<25');
ax[1].legend();
metro[metro.nom == "Ardennes"]
| code_insee | nom | DEPLONG | DEPLAT | taux_occupation_sae | R | |
|---|---|---|---|---|---|---|
| 31 | 08 | Ardennes | 4.640751 | 49.616226 | 22.4 | NaN |