Cube multidimensionnel - énoncé#
Links: notebook, html, PDF, python, slides, GitHub
Ce notebook aborde différentes solutions pour traiter les données qu’on représente plus volontiers en plusieurs dimensions. Le mot-clé associé est OLAP ou cube OLAP. Mondrian est une solution open source, cubes est écrit en python. Kylin propose ce service sur des données stockées sur Hadoop. L’objectif est ici de découvrir pas d’explorer ces solutions.
%matplotlib inline
import matplotlib.pyplot as plt
plt.style.use('ggplot')
import pyensae
from pyquickhelper.helpgen import NbImage
from jyquickhelper import add_notebook_menu
add_notebook_menu()
Populating the interactive namespace from numpy and matplotlib
Représentation#
Le module pandas manipule des tables et c’est la façon la plus commune de représenter les données. Lorsque les données sont multidimensionnelles, on distingue les coordonnées des valeurs :
NbImage("cube1.png")
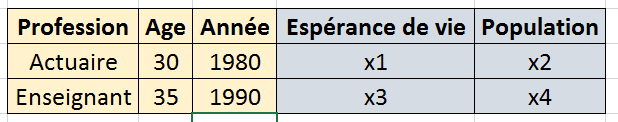
Dans cet exemple, il y a :
3 coordonnées : Age, Profession, Annéee
2 valeurs : Espérance de vie, Population
On peut représenter les donnés également comme ceci :
NbImage("cube2.png")
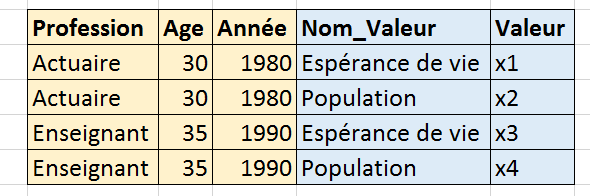
C’est assez simple. Prenons un exemple : table de mortalité de 1960 à 2010 qu’on récupère à l’aide de la fonction table_mortalite_euro_stat. C’est assez long (4-5 minutes) sur l’ensemble des données car elles doivent être prétraitées (voir la documentation de la fonction). Pour écouter, il faut utiliser le paramètre stop_at.
from actuariat_python.data import table_mortalite_euro_stat
table_mortalite_euro_stat()
'mortalite.txt'
import os
os.stat("mortalite.txt")
os.stat_result(st_mode=33206, st_ino=73183493945221205, st_dev=2797837379, st_nlink=1, st_uid=0, st_gid=0, st_size=104871002, st_atime=1445699900, st_mtime=1445699915, st_ctime=1445699900)
import pandas
df = pandas.read_csv("mortalite.txt", sep="\t", encoding="utf8", low_memory=False)
df.head()
| annee | valeur | age | age_num | indicateur | genre | pays | |
|---|---|---|---|---|---|---|---|
| 0 | 2009 | 0.00080 | Y01 | 1 | DEATHRATE | F | AM |
| 1 | 2008 | 0.00067 | Y01 | 1 | DEATHRATE | F | AM |
| 2 | 2007 | 0.00052 | Y01 | 1 | DEATHRATE | F | AM |
| 3 | 2006 | 0.00123 | Y01 | 1 | DEATHRATE | F | AM |
| 4 | 2013 | 0.00016 | Y01 | 1 | DEATHRATE | F | AT |
Cubes de données avec xarray#
création#
Cette réprésentation sous forme de table n’est pas toujours très lisible. Les informations sont souvent répétées et les données sont vraiment multidimensionnelles. Le module xarray propose de distinguer les coordonnées des valeurs pour proposer des manipulations plus intuitives. Le module propose un DataFrame multidimensionnelle DataSet.
from actuariat_python.data import table_mortalite_euro_stat
table_mortalite_euro_stat()
import pandas
df = pandas.read_csv("mortalite.txt", sep="\t", encoding="utf8", low_memory=False)
df.columns
Index(['annee', 'valeur', 'age', 'age_num', 'indicateur', 'genre', 'pays'], dtype='object')
Dans ce cas-ci, pour reprendre la temrinologie du module xarray, nous avons :
les dimensions : annee, age_num, pays, genre
les valeurs : une valeur par indicateur
On peut passer d’un DataFrame à un DataSet de la façon suivante :
les colonnes indéxées représentent les dimensions
les colonnes non indéxées sont les valeurs
On garde supprime les colonnes qui ne nous intéressent pas et les valeurs manquantes :
df2 = df[["annee", "age_num","indicateur","pays","genre","valeur"]].dropna().reset_index(drop=True)
df.columns, df2.columns
(Index(['annee', 'valeur', 'age', 'age_num', 'indicateur', 'genre', 'pays'], dtype='object'),
Index(['annee', 'age_num', 'indicateur', 'pays', 'genre', 'valeur'], dtype='object'))
df2.head()
| annee | age_num | indicateur | pays | genre | valeur | |
|---|---|---|---|---|---|---|
| 0 | 2009 | 1 | DEATHRATE | AM | F | 0.00080 |
| 1 | 2008 | 1 | DEATHRATE | AM | F | 0.00067 |
| 2 | 2007 | 1 | DEATHRATE | AM | F | 0.00052 |
| 3 | 2006 | 1 | DEATHRATE | AM | F | 0.00123 |
| 4 | 2013 | 1 | DEATHRATE | AT | F | 0.00016 |
df2["indicateur"] = df2["indicateur"].astype(str)
df2["genre"] = df2["genre"].astype(str)
df2["pays"] = df2["pays"].astype(str)
df2.dtypes
annee int64
age_num float64
indicateur object
pays object
genre object
valeur float64
dtype: object
L’opération complexe consiste à faire passer les valeurs de la colonne indicateur en tant que colonnes. C’est l’objet de la méthode pivot_table :
piv = df2.pivot_table(index=["annee", "age_num","pays","genre"],
columns=["indicateur"],
values="valeur")
piv.head()
| indicateur | DEATHRATE | LIFEXP | PROBDEATH | PROBSURV | PYLIVED | SURVIVORS | TOTPYLIVED | |||
|---|---|---|---|---|---|---|---|---|---|---|
| annee | age_num | pays | genre | |||||||
| 1960 | 1 | BE | F | 0.00159 | 73.7 | 0.00159 | 0.99841 | 97316 | 97393 | 7179465 |
| M | 0.00207 | 68.2 | 0.00207 | 0.99793 | 96420 | 96520 | 6580601 | |||
| T | 0.00184 | 70.9 | 0.00184 | 0.99816 | 96856 | 96945 | 6874529 | |||
| BG | F | 0.00652 | 73.2 | 0.00650 | 0.99350 | 95502 | 95813 | 7017023 | ||
| M | 0.00644 | 70.0 | 0.00642 | 0.99358 | 94801 | 95107 | 6653130 |
piv.dtypes
indicateur
DEATHRATE float64
LIFEXP float64
PROBDEATH float64
PROBSURV float64
PYLIVED float64
SURVIVORS float64
TOTPYLIVED float64
dtype: object
Les données sont maintenant prêtes à passer sous xarray :
import xarray
ds = xarray.Dataset.from_dataframe(piv)
ds
<xarray.Dataset>
Dimensions: (age_num: 84, annee: 54, genre: 3, pays: 54)
Coordinates:
* annee (annee) int64 1960 1961 1962 1963 1964 1965 1966 1967 1968 ...
* age_num (age_num) float64 1.0 2.0 3.0 4.0 5.0 6.0 7.0 8.0 9.0 10.0 ...
* pays (pays) object 'AM' 'AT' 'AZ' 'BE' 'BG' 'BY' 'CH' 'CY' 'CZ' ...
* genre (genre) object 'F' 'M' 'T'
Data variables:
DEATHRATE (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
LIFEXP (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBDEATH (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBSURV (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
SURVIVORS (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
TOTPYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
sélection#
Il est facile ensuite d’extraire les données d’un pays avec la méthode sel :
ds.sel(pays=["FR"])
<xarray.Dataset>
Dimensions: (age_num: 84, annee: 54, genre: 3, pays: 1)
Coordinates:
* annee (annee) int64 1960 1961 1962 1963 1964 1965 1966 1967 1968 ...
* age_num (age_num) float64 1.0 2.0 3.0 4.0 5.0 6.0 7.0 8.0 9.0 10.0 ...
* pays (pays) object 'FR'
* genre (genre) object 'F' 'M' 'T'
Data variables:
DEATHRATE (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
LIFEXP (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBDEATH (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBSURV (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
SURVIVORS (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
TOTPYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
Ou de plusieurs pays :
ds.sel(pays=["FR", "BE"])
<xarray.Dataset>
Dimensions: (age_num: 84, annee: 54, genre: 3, pays: 2)
Coordinates:
* annee (annee) int64 1960 1961 1962 1963 1964 1965 1966 1967 1968 ...
* age_num (age_num) float64 1.0 2.0 3.0 4.0 5.0 6.0 7.0 8.0 9.0 10.0 ...
* pays (pays) object 'FR' 'BE'
* genre (genre) object 'F' 'M' 'T'
Data variables:
DEATHRATE (annee, age_num, pays, genre) float64 nan nan nan 0.00159 ...
LIFEXP (annee, age_num, pays, genre) float64 nan nan nan 73.7 68.2 ...
PROBDEATH (annee, age_num, pays, genre) float64 nan nan nan 0.00159 ...
PROBSURV (annee, age_num, pays, genre) float64 nan nan nan 0.9984 ...
PYLIVED (annee, age_num, pays, genre) float64 nan nan nan 9.732e+04 ...
SURVIVORS (annee, age_num, pays, genre) float64 nan nan nan 9.739e+04 ...
TOTPYLIVED (annee, age_num, pays, genre) float64 nan nan nan 7.179e+06 ...
Ou plusieurs dimensions :
ds.sel(pays="FR", annee=2000)
<xarray.Dataset>
Dimensions: (age_num: 84, genre: 3)
Coordinates:
annee int64 2000
* age_num (age_num) float64 1.0 2.0 3.0 4.0 5.0 6.0 7.0 8.0 9.0 10.0 ...
pays object 'FR'
* genre (genre) object 'F' 'M' 'T'
Data variables:
DEATHRATE (age_num, genre) float64 0.00043 0.00046 0.00044 0.00021 ...
LIFEXP (age_num, genre) float64 82.3 74.7 78.5 81.3 73.7 77.6 80.3 ...
PROBDEATH (age_num, genre) float64 0.00043 0.00046 0.00044 0.00021 ...
PROBSURV (age_num, genre) float64 0.9996 0.9995 0.9996 0.9998 0.9997 ...
PYLIVED (age_num, genre) float64 9.957e+04 9.944e+04 9.951e+04 ...
SURVIVORS (age_num, genre) float64 9.959e+04 9.947e+04 9.953e+04 ...
TOTPYLIVED (age_num, genre) float64 8.196e+06 7.432e+06 7.817e+06 ...
Pour accéder à la série LIFEXP pour les hommes, les années 2000 et
2010, le pays FR, on fait la différence, puis on la récupère sous
forme de DataFrame :
(ds.sel(pays="FR", annee=2010, genre="T")["LIFEXP"] -
ds.sel(pays="FR", annee=2000, genre="T")["LIFEXP"]).to_dataframe().head()
| LIFEXP | genre | pays | |
|---|---|---|---|
| age_num | |||
| 1 | 2.6 | T | FR |
| 2 | 2.6 | T | FR |
| 3 | 2.6 | T | FR |
| 4 | 2.6 | T | FR |
| 5 | 2.6 | T | FR |
On a gagné presque deux ans et demi d’espérance de vie à la naissance en 10 ans.
(ds.sel(pays="FR", annee=2010, genre=["F","M"])["LIFEXP"] -
ds.sel(pays="UK", annee=2010, genre=["F","M"])["LIFEXP"]).to_dataframe().head()
| LIFEXP | annee | ||
|---|---|---|---|
| age_num | genre | ||
| 1 | F | 2.6 | 2010 |
| M | -0.3 | 2010 | |
| 2 | F | 2.7 | 2010 |
| M | -0.4 | 2010 | |
| 3 | F | 2.7 | 2010 |
Quelques graphes#
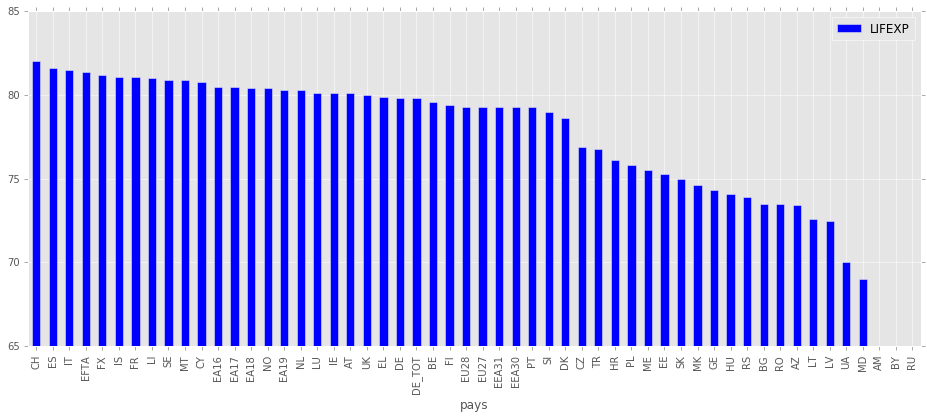
ds.sel(annee=2010,age_num=1,genre="T")["LIFEXP"].to_dataframe() \
.sort_values("LIFEXP", ascending=False) \
.plot(y="LIFEXP", kind="bar", figsize=(16,6), ylim=[65,85])
<matplotlib.axes._subplots.AxesSubplot at 0x1bf22320>
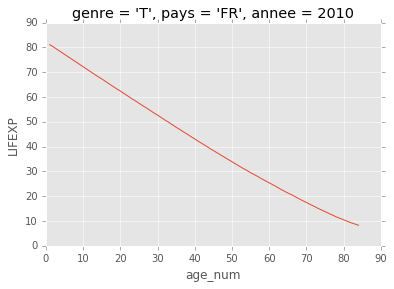
ds.sel(annee=2010,genre="T",pays="FR")["LIFEXP"].plot()
[<matplotlib.lines.Line2D at 0x1539ca58>]
Exercice 1 : que font les lignes suivantes ?#
On pourra s’aider des pages :
ds.assign(LIFEEXP_add = ds.LIFEXP-1)
<xarray.Dataset>
Dimensions: (age_num: 84, annee: 54, genre: 3, pays: 54)
Coordinates:
* annee (annee) int64 1960 1961 1962 1963 1964 1965 1966 1967 1968 ...
* age_num (age_num) float64 1.0 2.0 3.0 4.0 5.0 6.0 7.0 8.0 9.0 10.0 ...
* pays (pays) object 'AM' 'AT' 'AZ' 'BE' 'BG' 'BY' 'CH' 'CY' 'CZ' ...
* genre (genre) object 'F' 'M' 'T'
Data variables:
DEATHRATE (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
LIFEXP (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBDEATH (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBSURV (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
SURVIVORS (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
TOTPYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
LIFEEXP_add (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
meanp = ds.mean(dim="pays")
ds1, ds2 = xarray.align(ds, meanp, join='outer')
joined = ds1.assign(meanp = ds2["LIFEXP"])
joined.to_dataframe().head()
| DEATHRATE | LIFEXP | PROBDEATH | PROBSURV | PYLIVED | SURVIVORS | TOTPYLIVED | meanp | ||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| age_num | annee | genre | pays | ||||||||
| 1 | 1960 | F | AM | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 73.52 |
| AT | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 73.52 | |||
| AZ | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 73.52 | |||
| BE | 0.00159 | 73.7 | 0.00159 | 0.99841 | 97316 | 97393 | 7179465 | 73.52 | |||
| BG | 0.00652 | 73.2 | 0.00650 | 0.99350 | 95502 | 95813 | 7017023 | 73.52 |
Lire, écrire des datasets#
Le module xarray s’appuie sur le module netCDF4 qui lui-même est un wrapper poython de la libraire netCDF-c. Celle-ci est spécialisée dans la lecture et l’écriture de données scientifiques. Concrètement, ce n’est pas xarray qui s’en occupe mais netCDF4. Cela explique la syntaxe décrite par Serialization and IO :
ds.data_vars
Data variables:
DEATHRATE (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
LIFEXP (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBDEATH (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PROBSURV (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
PYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
SURVIVORS (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
TOTPYLIVED (annee, age_num, pays, genre) float64 nan nan nan nan nan ...
try:
ds.to_netcdf('mortalite.nc')
except ValueError as e:
# it breaks with pandas 0.17
# xarray has to be updated
print("l'écriture a échoué")
pass
l'écriture a échoué
C’est un format binaire plus efficace que le format texte :
import os
if os.path.exists("mortalite.nc"):
os.stat('mortalite.nc').st_size, os.stat('mortalite.txt').st_size
On relit :
if os.path.exists("mortalite.nc"):
ds_lu = xarray.open_dataset('mortalite.nc')
ds_lu
Le module xarray propose également de lire des données de plusieurs fichiers pour ne former qu’un seul dataset (voir Combining multiple files) :
pays = list(_.values for _ in ds["pays"])
pays[:5]
['AM', 'AT', 'AZ', 'BE', 'BG']
for p in pays[:5]:
print("enregistre", p)
d = ds.sel(pays=[p])
try:
d.to_netcdf("mortalite_pays_%s.nc" % p)
except ValueError:
print("l'écriture a échoué pour", p)
enregistre AM
l'écriture a échoué pour AM
enregistre AT
l'écriture a échoué pour AT
enregistre AZ
l'écriture a échoué pour AZ
enregistre BE
l'écriture a échoué pour BE
enregistre BG
l'écriture a échoué pour BG
On relit :
import os
if os.path.exists("mortalite_pays_AM.nc"):
ds_lu2 = xarray.open_mfdataset('mortalite_pays*.nc')
ds_lu2
A quoi ça sert ?
Lorsqu’on travaille avec des gros fichiers de données, on cherche à retarder le plus possible l’exécution d’un calcul, on manipule les données sans les charger en mémoire. On appelle ceci évaluation paresseuse ou lazy evaluation en anglais. xarray et dask permettent de faire cela (voir Using dask with xarray). La logique qu’on suit est alors assez proche de la programmation fonctionnelle.